7 - Seleção e filtragem
As principais formas de seleção de um ou mais valores de um dataframe df do tipo DataFrame são os métodos:
- df.loc[]
- df.iloc[]
- df.at[]
- df.iat[]
Um subconjunto de dados do dataframe, seja por seleção de linhas, colunas ou ambas, é denominado de fatia ou slice.
A principal diferença entre loc/at e iloc/iat é a seguinte:
- loc[] e at[] são baseados em rótulos ou nomes das linhas ou colunas
- iloc[] e iat[] são baseados nos índices numéricos (mesmo que tenham nomes) sempre com base 0.
at[] e loc[]
- df.at[rot_lin, rot_col]
- df.loc[rot_lin,rot_col]
iat[] e iloc[]
- df.iat[pos_lin, pos_col]
- df.iloc[pos_lin,pos_col]
Tipos retornados
Os tipos retornados podem ser:
| Simbolo | Descrição |
|---|---|
| O | O elemento da m-ésima linha, n-ésima coluna. |
| S | Linha (tipo Series). |
| D | Tabela (tipo DataFrame). |
Nas operações df.at[] e df.iat[] os índices de linhas e colunas se referem a posições.
| Operações df.iat e df.at | Tipo | Retorno |
|---|---|---|
| df.iat[m,n] | O | elemento da m-ésima linha, n-ésima coluna |
| df.at[rot_lin,rot_col] | O | elemento linha/coluna relativas aos labels rot_lin/rot_col |
Nas operações df.iloc[] os índices de linhas e colunas se referem a posições.
| Operações df.iloc | Tipo | Retorno |
|---|---|---|
| df.iloc[n] | S | n-ésima linha |
| df.iloc[[n]] | D | n-ésima linha |
| df.iloc[-n] | S | n-ésima linha, contando do final |
| df.iloc[i,j:, n] | S | linhas i, até j (excluindo), coluna n |
| df.iloc[[i,j,k]:[m,n,o]] | D | linhas i, j, k, colunas m, n, o |
| df.iloc[:, n] | S | n-ésima coluna |
| df.iloc[:, [n]] | D | n-ésima coluna |
| df.iloc[:,-1] | S | última coluna |
| df.iloc[i:j,m:n] | D | linhas i até j (excluindo), colunas m até n (inclusive) |
Nas operações df.loc[] os índices de linhas e colunas se referem aos rótulos.
| Operações df.loc | Tipo | Retorno |
|---|---|---|
| df.loc[n] | S | linha de índice n |
| df.loc[[n]] | D | linha de índice n |
| df.loc[:] | D | todas as linhas e colunas |
| df.loc[:, 'col'] | S | todas as linhas, coluna 'col' |
| df.loc[:, ['col']] | D | todas as linhas, coluna 'col' |
| df.loc[:, ['col1', 'col2']] | D | todas as linhas, colunas 'col1' e 'col2' |
| df.loc[i:j, ['col1', 'col2']] | D | linhas com índices de i até i (inclusive), colunas 'col1' e 'col2' |
| df.loc[[i,j,k] , ['col1', 'col2']] | D | linhas com índices i, j, k, colunas 'col1' e 'col2' |
| df.loc[i:j, 'col1':'coln'] | D | linhas com índices i até j (inclusive), colunas 'col1' até 'coln' (inclusive) |
Atalhos e equivalentes:
| Atalho | Tipo | Equivalente |
|---|---|---|
| df['col1'] ou df.col1 | S | df.loc[:,'col1'] |
| df[['col1','col2']] | D | df.loc[:,['col1','col2']] |
Em todos esses métodos uma exceção de KeyError é levantada se um índice ou label não existir no dataframe.
Se o index da linha coincidir com sua posição então df.loc[n] e df.iloc[n] serão as mesmas linhas.
Isso nem sempre é verdade, como se verá abaixo com o reordenamento das linhas.
São incorretas as sintaxes, como a seguir, de loc[] usando n numérico, pois devem ser utilizados rótulos.
n = 1
df.loc[-n]
df.loc[:,n]
df.loc[:,[n]]7.1 - Exemplos de consultas e seleções.
Importe a biblioteca Pandas.
import pandas as pdO dataframe dfPaises é carregado para fazer consultas e seleções.
import numpy as np
dfPaises = pd.read_csv('dados_demograficos_paises.csv')
print(dfPaises)7.1.1 - df.iloc[]
Primeiro, usando a função df.iloc() para selecionar uma linha.
Lembramos que a contagem de índices sempre se inicia em 0.
Observação: dfPaises.iloc[0] retorna uma série da classe Series e dfPaises.iloc[[0]] retorna um dataframe da classe DataFrame.
Primeira linha, pelo índice:
aux = dfPaises.iloc[0]
print("*** Tipo: ", type(aux), end="\n\n")
print(aux)Primeira linha, pelo índice:
aux = dfPaises.iloc[[0]]
print("*** Tipo: ", type(aux), end="\n\n")
print(aux)Última linha, pelo índice:
print(print(dfPaises.iloc[[-1]]))Linhas 15 até 20 (excluindo), colunas 2 até 5 (excluindo):
print(dfPaises.iloc[15:20, 2:5])Linhas 1, 3, 5 e colunas 2, 5:
print(dfPaises.iloc[[1,3,5],[2,5]])Linhas 1, 3, 5, última coluna:
print(dfPaises.iloc[[1,3,5],-1])Todas as linhas, coluna 3:
print(dfPaises.iloc[:, [3]].head())Linhas 0, 3, 6, 24; colunas 0, 3, 5:
print(dfPaises.iloc[[0,3,6,24], [0,3,5]])A seleção das linhas nos dois métodos é diferente. Em df.loc[m,n] linhas com labels de m até n (inclusive) são selecionadas. Em df.iloc[m,n] são selecionadas linhas com índices (numéricos) de m até n (excluindo).
iloc[m,n] exibe linhas m até n (excluindo):
print(dfPaises.iloc[1:2])7.1.2 - df.loc[]
loc[m:n] exibe linhas m até n (inclusive):
print(dfPaises.loc[1:2])Os próximos testes são feitos com df.loc(), que deve receber os labels como índices.
Todas as linhas, só colunas 'ano' e 'populacao' (limitadas por head()):
print(dfPaises.loc[:,['ano','populacao']].head())Linhas 3 até 6 (inclusive), só colunas 'ano' e 'expVida':
print(dfPaises.loc[3:6,['ano', 'expVida']])Todas as linhas, só colunas 'ano' (restritas por head()):
print(dfPaises.loc[:, 'ano'].head())7.1.3 - Métodos df.loc, df.iloc, df.at e df.iat
Para explorar um pouco mais a diferença no uso de df.loc e df.iloc vamos criar um dataframe bem simples e sem valores nulos.
dic = {
'João' : {'Prova 1': 5.4, 'Prova 2': 6.2, 'Prova 3': 7.9},
'Maria' : {'Prova 1': 8.5, 'Prova 2': 9.7, 'Prova 3': 6.6},
'Rosana' : {'Prova 1': 5.0, 'Prova 2': 7.0, 'Prova 3': 4.3}
}
dfNotas = pd.DataFrame(dic)
print(dfNotas)df.loc e df.at usa labels de linhas e colunas.
df.iloc e df.iat usa números (índices) de linhas e colunas.
Nos comentários listamos seleções usando df.iloc para se obter o mesmo retorno.
aux = dfNotas.loc['Prova 1','Rosana']
print(type(aux))
print(aux) # dfNotas.iloc[0,2]aux = dfNotas.loc['Prova 1']
print(type(aux))
print(aux) # dfNotas.iloc[0]aux = dfNotas.loc[['Prova 1']]
print(type(aux))
print(aux) # dfNotas.iloc[[0]]aux = dfNotas.loc[['Prova 1','Prova 2']]
print(type(aux))
print(aux) # dfNotas.iloc[0:2]aux = dfNotas.loc['Prova 1':'Prova 3']
print(type(aux))
print(aux) # dfNotas.iloc[0:3]aux = dfNotas.loc[['Prova 1'],['Maria','Rosana']]
print(type(aux))
print(aux) # dfNotas.iloc[[0],[1,2]]aux = dfNotas.loc['Prova 1':'Prova 3', 'João':'Rosana']
print(type(aux))
print(aux) # dfNotas.iloc[0:3,0:3] (D)aux = dfNotas.loc[:,['Rosana']]
print(type(aux))
print(aux) # dfNotas.iloc[:,[2]] (S)Observe que em dfNotas.iloc[0:3,0:3] são selecionadas as linhas de índices 0, 1 e 2 e colunas 0, 1 e 2.
Análogos à df.loc e df.iloc temos, respectivamente, df.at[rot_lin, rot_col] e df.iat[m,n] onde rot_lin, rot_col se referem aos labels e m, n aos índices das linhas/colunas. Ambos recebem um par e retornam um único valor do df. Quando aplicados em uma Series iat e at recebem um único índice/label localizador de posição.
7.1.4 - df.at[] e df.iat[]
print(dfNotas.iat[0,2]) # dfNotas.at['Prova 1', 'Rosana']
print(dfNotas.iat[2,1]) # dfNotas.at['Prova 3', 'Maria']
print(dfNotas.iloc[0].iat[1]) # dfNotas.loc['Prova 1'].iat[1]
print(dfNotas.at['Prova 1', 'Rosana']) # dfNotas.iat[0,2]
print(dfNotas.loc['Prova 1'].at['Maria']) # o mesmo que dfNotas.loc['Prova 1'].iat[1]7.1.5 - df.loc[] e df.iloc[] com slices
Nenhuma das duas formas de seleção de uma slice (.loc ou .iloc) copiam um dataframe por referência, como ocorre com numPy.ndarrays.
Por exemplo, df = dfNotas.iloc[:,[2]] é uma cópia da 3ª coluna, e não uma referência ou view.
Ela pode ser alterada sem que o dataframe original seja modificado. Se um novo valor for atribuído ao slice diretamente, no entanto, o dataframe fica alterado.
df = dfNotas.iloc[:,[2]]
df.Rosana = 10
print(df)
print(dfNotas)No entanto se o slice receber atribuição direta o dataframe fica alterado
dfNotas.iloc[:,[2]] = 10
print(dfNotas)Para inserir valores diferentes outro dataframe deve ser atribuído ao slice.
dic = {'Rosana': {'Prova 1': 8.5, 'Prova 2': 7.9, 'Prova 3': 10}}
dfLuna = pd.DataFrame(dic)
dfNotas.iloc[:,[2]] = dfLuna
print(dfNotas)Alternativamente, um np.array com shape apropriado pode ser atribuído ao slice.
arrLuna = np.array([2.3, 4.5, 5.6]).reshape(3,1)
dfNotas.iloc[:,[2]] = arrLuna
print(dfNotas)Na atribuição dfNotas.iloc[:,[2]] = 10 houve o broadcasting de 10 para uma forma compatível com o slice.
Para que a atribuição seja bem sucedida, sem necessidade de broadcasting, um objeto de mesmo formato deve ser atribuído.
No caso dfNotas.iloc[:,[2]].shape = dfLuna.shape = (3, 1) (3 linhas, 1 coluna)
O mesmo ocorre com a atribuição de um array do numpy.
7.2 - Manipulando linhas e colunas
Um array booleano pode ser passado como índice de um dataframe.
Apenas as linhas correspondentes ao índice True são exibidas.
Alguns métodos de string estão disponíveis para testes em campos, como df['campo'].str.startswith('str') e df['campo'].str.endswith('str') (começa e termina com).
O teste df['campo'].isin(['str1', 'str2']) retorna um array booleano com True para cada linha que contém um dos valores passados.
Para os exemplos usamos o dataframe dfPaises.
Seleção por array booleano:
print(dfPaises.loc[dfPaises['ano'] == 2016].head(3))Quais os paises tem nome começados com 'Es':
print(dfPaises.loc[dfPaises['pais'].str.startswith('Es')]['pais'].unique())Quais os paises tem nome terminados em 'ia':
print(dfPaises.loc[dfPaises['pais'].str.endswith('ia')]['pais'].unique())Quantas linhas se referem à 'Europa' e 'África':
print(dfPaises.loc[dfPaises['continente'].isin(['Europa', 'África'])].shape[0])7.3 - Seleção por array booleano com múltiplas condições
Os conectores lógicos são:
| Símbolo | Operador | significa |
|---|---|---|
| & | and | e |
| | | or | ou |
| ~ | not | negação |
print(dfPaises.loc[(dfPaises['continente']=='África') & (dfPaises['ano']==2016)].head(4))Paises e anos com população < 7000000 ou expectativa de vida > 82
print(dfPaises.loc[(dfPaises['populacao'] < 7000000) | (dfPaises['expVida'] > 82)][['ano','pais']])O método arr.unique() acima foi aplicado para ver quais os países satisfazem as condições, sem repetições.
arr.shape é uma tupla (número linhas, número colunas).
Os últimos exemplos fazem testes compostos usando os operadores & (and, e lógico) e | (or, ou lógico).
Se nenhum campo for submetido ao teste lógico todos os valores do dataframe são usados.
O mesmo ocorre com a aplicação de uma função, como mostrado para uma função lambda.
7.4 - Testes das funções loc e iloc
Criamos o dataframe dfNotas com 3 linhas e 3 colunas.
dic = {
'João' : {'Prova 1': 5.4, 'Prova 2': 6.2, 'Prova 3': 7.9},
'Maria' : {'Prova 1': 8.5, 'Prova 2': 9.7, 'Prova 3': 6.6},
'Rosana' : {'Prova 1': 5.0, 'Prova 2': 7.0, 'Prova 3': 4.3}
}
dfNotas = pd.DataFrame(dic)
print(dfNotas)O teste retorna um df com o mesmo shape que dfNotas
print(dfNotas > 6)Os campos do df são filtrados pelo df booleano
print(dfNotas[dfNotas > 6])7.4.1 - Funções lambda
Uma função pode ser aplicada sobre elementos de uma coluna específica ou sobre todas as colunas.
Veremos posteriormente detalhes sobre o uso de df.apply().
print(dfNotas)Uma função aplicada à todos os elementos do df
print(dfNotas.apply(lambda x: x**2))Funções lambda que retornam valores booleanos podem ser usadas para filtragem dos campos de um df.
No exemplo dfPaises['pais'].apply(lambda x: len(x)) == 4 retorna True para as linhas onde o campo pais tem comprimento de 4 letras.
print(dfPaises.loc[dfPaises['pais'].apply(lambda x: len(x)) == 4].head(2))São os países com nomes de 4 letras:
paises = dfPaises.loc[dfPaises['pais'].apply(lambda x: len(x)) == 4]['pais']
print(paises.head(2))
print(set(paises)) # o mesmo que paises.unique()O seletor pode ser composto de mais testes, ligados pelos operadores lógicos & e |.
Paises/anos com nomes compostos por mais de 2 palavras e população acima de 50 milhões
print(dfPaises.loc[
(dfPaises['pais'].apply(lambda x: len(x.split(' '))) > 1) &
(dfPaises['populacao'] > 50_000_000)])7.5 - Ordenamento com Sort
Para ordenar um dataframe podemos usar o método sort, com a seguinte sintaxe:
df.sort_values(by=['campo'], axis=0, ascending=True, inplace=False)Onde:
- by: pode ser uma string ou lista com o nome ou nomes dos campos, na prioridade de ordenamento
- axis{0 ou 'index', 1 ou 'columns'}: indica o eixo a ordenar, default 0
- ascending=True/False: se ordenamento é crescente/decrescente
Existem vários outros parâmetros para o controle de ordenamentos, como pode ser lido no API de referência do pandas.
Muitas informações importantes sobre um conjunto de dados podem ser obtidas apenas pela inspecção dos dados.
Por exemplo, podemos encontrar respostas para:
- que país do mundo teve, em qualquer ano, o PIB percapita mais elevado?
- no ano de 2018 (o último de nossa lista), quais são os 5 países com maior população, e quais são os 5 com PIB mais baixo, no mundo?
- no ano de 2016, quantos países no mundo tinham PIB percapita acima e abaixo da média?
Encontramos o maior pib percapita e a linha que corresponde a ele:
dfMax = dfPaises[dfPaises['pibPercap']==dfPaises['pibPercap'].max()]
print(dfMax)Para formatar uma resposta amigável:
ano = dfMax['ano'].values[0]
pais = dfMax['pais'].values[0]
pibP = dfMax['pibPercap'].values[0]
print('O PIB percapita máximo foi de {} e ocorreu no {} em {}.'.format(pibP, pais, ano))Ordenando em ordem decrescente
print(dfPaises.sort_values(by=['pibPercap'], ascending=False).iloc[[0]])Observe que dfMax['ano'] é do tipo Series que, se exposta diretamente, não contém apenas o ano.
Por isso extraimos dele o valor, 1º campo: dfMax['ano'].value[0]. Idem para pais e pibPercap.
Claro que podemos também ordenar o dataframe em ordem descrecente no campo pibPercap e pegar apenas a 1ª linha.
df.iloc[[0]] foi usado para pegar a 1ª linha, cujo índice é 853.
A mesma linha seria retornada com df.loc[[853]], o que mostra, mais uma vez, a diferença entre df.loc e df.iloc.
Para encontrar os 5 países com maior população em 2018 usamos a mesma técnica de ordenamento.
Primeiro filtramos pelo ano = 2018, ordenamos por população, ordem inversa, e pegamos os 5 primeiros.
Para exibir o resultado podemos transformar o dataframe em string, sem os índices.
Encontrar os 5 países com maior população em 2018, e os 5 com menor PIB.
Os 5 países com maior população em 2018:
popMax = dfPaises[dfPaises['ano']==2018].sort_values(by=['populacao'], ascending=False).head()
print(popMax[['pais','populacao']].to_string(index=False))A seguir os 5 países com menor PIB em 2018.
criamos um dataframe apenas do ano 2018 e acrescentamos o campo pib = pibPercap * populacao.
df2007 = dfPaises[dfPaises['ano']==2018]
df2007['pib'] = df2007['pibPercap'] * df2007['populacao']São os países com menor pib em 2018:
print(df2007.sort_values(by=['pib']).head()['pais'])Se não precisamos mais do df, podemos apagá-lo.
del df2007Para saber quantos países tem PIB percapita acima e abaixo da média em 2016 primeiro encontramos essa média.
Depois selecionamos as linhas que satisfazem com pibPercap >= media e pibPercap < media.
Para saber quantas linhas restaram contamos, por exemplo, quantos elementos existem em seu index.
Média do pibPercap em 2016 (um escalar):
media2016 = dfPaises[dfPaises.ano==2016]['pibPercap'].mean()
acima = dfPaises[(dfPaises.ano==2016) & (dfPaises.pibPercap >= media2016)].index.size
abaixo = dfPaises[(dfPaises.ano==2016) & (dfPaises.pibPercap < media2016)].index.size
print('Dos {} países, {} tem PIB percapita acima da média, {} abaixo da média.'.format(acima+abaixo, acima, abaixo))Obtenção e análise de um slice : Brasil
Em diversas circunstâncias queremos fazer análise de apenas um slice da dataframe geral.
Além de simplificar o conjunto de campos podemos conseguir com isso um uso menor de espaço em memória e maior velocidade de processamento.
Podemos, por ex., obter um dataframe separado apenas com a os dados referentes ao Brasil.
Passando como índice o array booleano dfPaises['pais'] == 'Brasil' apenas as linhas relativas a esse país serão retornadas.
dfBrasil = dfPaises[dfPaises['pais'] == 'Brasil'][['ano', 'expVida', 'populacao', 'pibPercap']]
print(dfBrasil.head())O dataframe dfBrasil tem os mesmos índices que aos do segmento de dfPaises, de onde ele foi retirado.
Para restabelecer esses índices usamos dataFrame.reset_index().
Se utilizado com o parâmetro drop=True o índice antigo é excluído (e perdido), caso contrário é copiado como uma coluna do df.
Para atribuir um nome para o índice usamos df.index.rename('novoNome', inplace=True).
Os índices iniciais são:
print(dfBrasil.index)Resetamos os índices, abandonando a coluna de índices iniciais:
dfBrasil.reset_index(drop=True, inplace=True)Novos índices:
print(dfBrasil.index)Agora podemos renomear o índice:
dfBrasil.index.rename('id', inplace=True)Podemos usar um campo qualquer como index, com qualquer dtype. No caso abaixo usamos o campo ano como índice.
Vamos usar o campo ano como index:
dfBrasil.set_index('ano', inplace=True)
print(dfBrasil.head(3))Agora os índices passam a ser o ano:
print("tipo Series: ")
print(dfBrasil.loc[2017]) # série
print("tipo DataFrame: ")
print(dfBrasil.loc[[2017]]) # dataframePara restaurar a coluna ano copiamos o índice para essa coluna e restauramos o índice.
Restauramos a coluna ano
dfBrasil['ano'] = dfBrasil.index
dfBrasil.reset_index(drop=True, inplace=True)
print(dfBrasil.head(3))Linhas podem ser inseridas de várias formas.
Um delas consiste em criar novos dataframes com as linhas a inserir e concatenar como a dataframe inicial.
Para isso usamos pandas.concat(): pd.concat([dfInicio, dfFinal]).
Vamos inserir linhas com dados fictícios, apenas para efeito de aprendizado.
colunas = ['expVida','populacao','pibPercap','ano'] # nomes das colunas, na ordem dos dados
valores1 = [48.0,45000000,2000.0,1951] # valores a inserir no ínicio (ano 1951)
valores2 = [75.0, 200000000, 9500.0, 2008] # valores a inserir no final (ano 2008)
dfP = pd.DataFrame([valores1], columns=colunas) # df a inserir no ínicio
dfU = pd.DataFrame([valores2], columns=colunas) # df a inserir no final
dfBrasil = pd.concat([dfP, dfBrasil]) # 1ª linha + dfBrasil
dfBrasil = pd.concat([dfBrasil, dfU]) # dfBrasil + última linhaAgora a 1ª linha é:
print(dfBrasil.iloc[[0]])A última linha é:
print(dfBrasil.iloc[[-1]])Como os índices ficaram duplicados e desordenados fazemos um reordenamento:
dfBrasil.reset_index(drop=True, inplace=True)
print(dfBrasil)Como essas linhas não contém dados corretos, vamos apagá-las.
Para usamos df.drop(linha, axis=0, inplace = True), onde linha é o label ( que pode não ser numérico), da linha ou seu índice (numérico).
Várias linhas podem ser apagadas com df.drop([linha0,...,linhan], axis=0, inplace = True).
Apagar linhas 0 e 2: axis = 0 se refere às linhas:
dfBrasil.drop([0,2], axis=0, inplace = True)
print(dfBrasil)Para reordenar os índices:
dfBrasil.reset_index(drop=True, inplace=True)
print(dfBrasil)Recolocar a coluna 'ano' no início:
dfBrasil = dfBrasil[['ano', 'expVida', 'populacao', 'pibPercap']]
print(dfBrasil)O estado do dataframe agora é:
print(dfBrasil)Vamos inserir uma coluna, atribuindo a ela um escalar (um valor único).
Aqui ocorre, como nas Series, o broadcasting, onde o escalar é transformado em uma Series de tamanho apropriado antes de ser inserido na nova coluna.
Todas as linhas terão o valor 42 no campo “novoCampo”.
Em seguida alteramos o valor dessa coluna em uma linha específica, usando df.loc(númeroLinha, nomeColuna) ou df.iloc(numeroLinha, numeroColuna).
Depois, como essa é uma coluna indesejada, nos a apagamos usando df.drop('nomeColuna', axis=1, inplace=True).
dfBrasil['novoCampo'] = 42
print(dfBrasil.head(3))Alteramos o 'novoCampo' na linha 1 (usando loc) e a coluna 4 ('novoCampo') na linha 2 (usando iloc, fornecendo o índice):» dfBrasil.loc[1,'novoCampo'] = 123456
dfBrasil.iloc[2,4] = 22222
print(dfBrasil.head(3))Apagamos essa coluna com drop
dfBrasil.drop('novoCampo', axis=1, inplace=True)O dataframe fica como no início.
Um campo pode ser inserido como resultado de operações entre outros campos.
No caso abaixo criamos uma coluna pib que é o produto das colunas populacao × pibPercap.
O resultado é aplicado, em cada linha, à nova coluna, em notação científica.
Na 1ª linha pib = 1.193716 × 1011.
Outra coluna marca a passagem de quando a expectativa de vida do brasileiro ultrapassa os 60 anos.
dfBrasil.loc[:,'pib'] = dfBrasil['pibPercap'] * dfBrasil['populacao']
print(dfBrasil.head(4))Inserindo coluna 'acima60':
dfBrasil.loc[:,'acima60'] = dfBrasil['expVida'] > 60
print(dfBrasil.loc[3:6,['ano','expVida','acima60']])Agora podemos usar essa coluna para selecionar linhas.
Todas as linhas com expVida > 60 (dfBrasil['expVida'] > 60 é uma série boleana) são exibidas (output omitido):
print(dfBrasil[dfBrasil['acima60']])As colunas podem ser removidas (para ficarmos com o dataframe original):
dfBrasil.drop(['acima60', 'pib'], axis=1, inplace=True)
print(dfBrasil)7.5.1 - Objetos de índices
Em um dataframe, assim como nas Series, a informação relativa aos índices e seus nomes (labels ), assim como os nomes dos eixos, são armazenados em objetos Index (índice).
O objeto Index é imutável (não pode ser alterado após a construção).
pdSerie = pd.Series(range(4), index=['a1', 'a2', 'a3', 'a4'])
index = pdSerie.index
print(index)o índice é uma sequência (pode ser lido em slices):
print(index[2])
print(index[2:])O index é imutável:
index[0] = 'A'Índices não fornecidos são preenchidos como um range:
print(pd.Series(range(4)).index)No exemplo abaixo construimos primeiro um objeto Index usando pandas.Index(lista).
Em seguida construimos uma Series usando esse index, contendo como valores as distâncias dos planeta até o Sol, em unidaddes astronômicas (UA).
Uma UA é a distância média da Terra ao Sol. 1 UA ≈ 149,6 × 109 m.
Com a Series inicializamos um dataframe com o mesmo index.
Objeto index
labels = pd.Index(np.array(['mercurio', 'venus', 'terra']))
print(labels)Série construída com esse index:
planetas = pd.Series([0.387, 0.723, 1], index=labels)
print(planetas)O index da Series é o mesmo objeto que labels:
print(planetas.index is labels)Essa Series pode ser usada para construir um dataframe:
dfPlanetas = pd.DataFrame(planetas)
print(dfPlanetas)O index do dataframe é o mesmo que o da Series:
print(dfPlanetas.index is labels)Alteramos o nome da coluna:
dfPlanetas.rename(columns={0:'distancia'}, inplace=True)
print(dfPlanetas)Podemos inserir uma coluna, por exemplo, relativa ao diâmetro dos planetas (comparados ao diâmetro da Terra), atribuindo valores à uma nova coluna de nome 'diametro'. O objeto atribuído deve ter o mesmo shape (ou passar por broadcasting). Alterar a ordem das colunas, o que pode ser feito com df.reindex(listaColunas), altera todo o dataframe (embora não inplace). O objeto retornado se ajusta de acordo com os índices fornecidos.
dfPlanetas['diametro'] = pd.Series([0.382, 0.949, 1], index=labels)
print(dfPlanetas)As colunas estão em um objeto Index:
print(dfPlanetas.columns)print(type(dfPlanetas.columns))print('distancia' in dfPlanetas.columns)Podemos alterar a ordem das colunas com reindex:
print(dfPlanetas.reindex(['venus','terra','mercurio']))Podemos ordenar os índices para ordenar o dataframe:
idx = dfPlanetas.index
idx = idx.sort_values()
print(idx)print(dfPlanetas.reindex(idx))Diferentes de um conjunto (set) objetos Index podem ter índices repetidos.
Se índices inseridos não correspondem à dados existentes estes são preenchidos com NaN.
Os parâmetros method='bfill' (ou “ffill”) forçam as colunas (ou linhas) com NaN a serem preenchidos com valores das colunas (ou linhas) anteriores ou posteriores.
Claro que reindexações podem ser também obtidas com df.loc e df.iloc.
Indices de linhas repetidos:
duplicados = pd.Index(['mercurio', 'venus', 'terra', 'mercurio', 'marte'])
print(duplicados)
print(dfPlanetas.reindex(duplicados)) # default é axis = 0Indices de colunas repetidos:
duplicados = pd.Index(['distancia', 'diametro', 'diametro', 'distancia', 'massa'])
print(dfPlanetas.reindex(duplicados, axis=1)) # sobre colunasO método reindex aceita um parâmetro method que pode ser usado para preencher valores faltantes com valores de colunas anteriores ou posteriores.
O argumento method='bfill' lê valor da coluna anterior:
print(dfPlanetas.reindex(duplicados, axis=1, method='bfill'))O argumento method='ffill' lê valor da coluna posterior.
Reindexação com loc:
nCol = pd.Index(['diametro', 'distancia'])
print(dfPlanetas.loc[['venus','terra'], ['diametro', 'distancia']])nCol pode ser uma lista: nCol = ['diametro', 'distancia']
De posse dos índices das linhas e colunas qualquer uma delas pode ser apagada com df.drop(lista, axis).
As operações retornam o dataframe modificado, sem alterar o original, a menos que seja marcado o parâmetro inplace=True.
Nesse caso os dados removidos serão perdidos.
print(dfPlanetas)Apagando linhas (axis = 0 é default):
print(dfPlanetas.drop(['venus', 'mercurio']))Apagando colunas:
print(dfPlanetas.drop(['distancia'], axis=1))Os seguintes argumentos são usados com reindex
| Argumento | descrição |
|---|---|
| index | Index ou sequência a ser usada como index, |
| method | forma de interpolação: 'ffill' preenche com valor posterior, 'bfill' com valor anterior, |
| fill_value | valor a usar quando dados não existentes são introduzidos por reindexing (ao invés de NaN), |
| limit | quando preenchendo com valor anterior ou posterior, intervalo máximo a preencher (em número de elementos), |
| tolerance | quando preenchendo com valor anterior ou posterior, intervalo máximo a preencher para valores inexatos (em distância numérica), |
| level | combina Index simples no caso de MultiIndex; caso contrário seleciona subset, |
| copy | se True, copia dados mesmo que novo índice seja equivalente ao índice antigo; se False, não copia dados quando índices são equivalentes. |
Métodos e propriedades de Index
| Método | descrição |
|---|---|
| append() | concatena outro objeto Index objects, gerando novo Index |
| difference() | calcula a diferença de conjunto como um Index |
| intersection() | calcula intersecção de conjunto |
| union() | calcula união de conjunto |
| isin() | retorna array booleano indicando se cada valor está na coleção passada |
| delete() | apaga índice, recalculando Index |
| drop() | apaga índices passados, recalculando Index |
| insert() | insere índice, recalculando Index |
| is_monotonic | retorna True se indices crescem de modo monotônico |
| is_unique | returns True se não existem valores duplicados no Index |
| unique | retorna índices sem repetições |
7.6 - Exemplo com dados de exercícios físicos
Pandas usa o método plot() do Pyplot do pacote Matplotlib para criar diagramas.
Importe os módulos necessários para os exemplos a seguir: o pandas e o pyplot do Matplotlib.
import pandas as pd
import matplotlib as mpl
import matplotlib.pyplot as plt
import seaborn as snsmpl.rcParams['figure.figsize'] = [12,9]
mpl.rcParams['figure.dpi'] = 80
mpl.rcParams['figure.titlesize'] = 32
mpl.rcParams['axes.titlesize'] = 24
mpl.rcParams['axes.labelsize'] = 20
mpl.rcParams['axes.xmargin'] = 0.01
mpl.rcParams['axes.ymargin'] = 0.01
mpl.rcParams['xtick.labelsize'] = 14
mpl.rcParams['ytick.labelsize'] = 14
mpl.rcParams["figure.subplot.left"] = 0.1
mpl.rcParams["figure.subplot.bottom"] = 0.07
mpl.rcParams["figure.subplot.right"] = 0.97
mpl.rcParams["figure.subplot.top"] = 0.9
mpl.rcParams['font.size'] = 10
mpl.rcParams['savefig.dpi'] = 100Os exemplos sobre os dados de exercícios físicos usam os do arquivo 'exercicios_fisicos.csv' no formato CSV.
Carregue o arquivo "exercicios_fisicos.csv" no formato CSV usando o método read_csv().
Baixar exercicios_fisicos.csv ou abrir exercicios-fisicos.csv
df = pd.read_csv('exercicios-fisicos.csv')
df_len = len(df)Imprima o cabeçalho do dataframe para relembrar. O completo se encontra em "Correlações".
print(df.head(5))Selecione as linhas com duração de 30 minutos.
df_aux = df[(df['Pulsomax'] >= 145)]
print(df_aux)Gráfico da seleção anterior.
df_aux.plot()
fig, axes = plt.subplots(nrows=1, ncols=3)
fig.suptitle('Pulso, Pulso máximo e calorias / 30 minutos')
fig.tight_layout(pad=3.0)
fig.subplots_adjust(left=0.1, top=0.7, right=0.9, bottom=0.1, wspace=0.2, hspace=0.5)
df_aux['Duracao'].plot(ax=axes[0],title='Duração (minutos)')
df_aux['Pulso'].plot(ax=axes[1],title='Pulso')
df_aux['Calorias'].plot(ax=axes[2],title='Calorias')
plt.show()
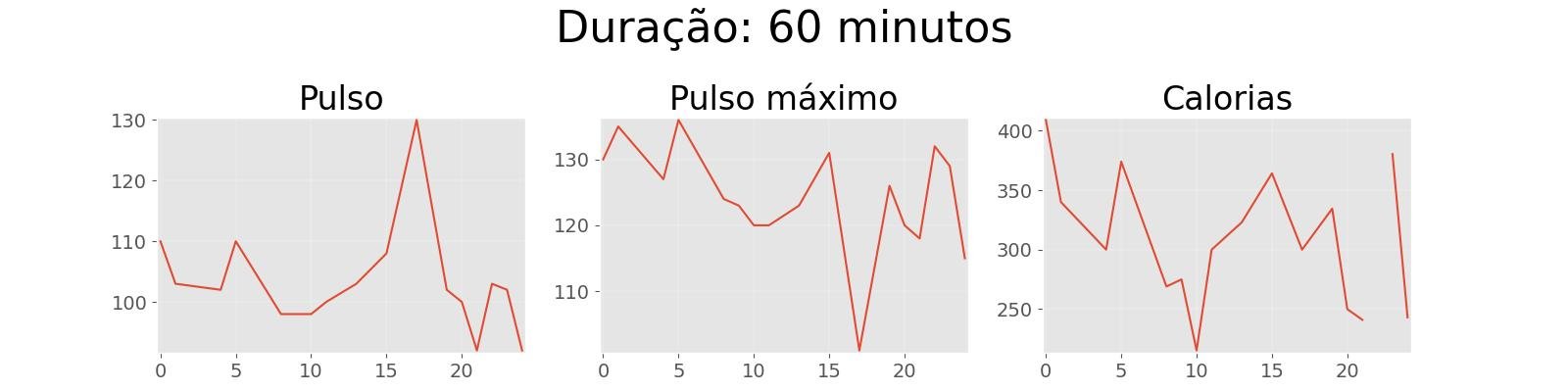
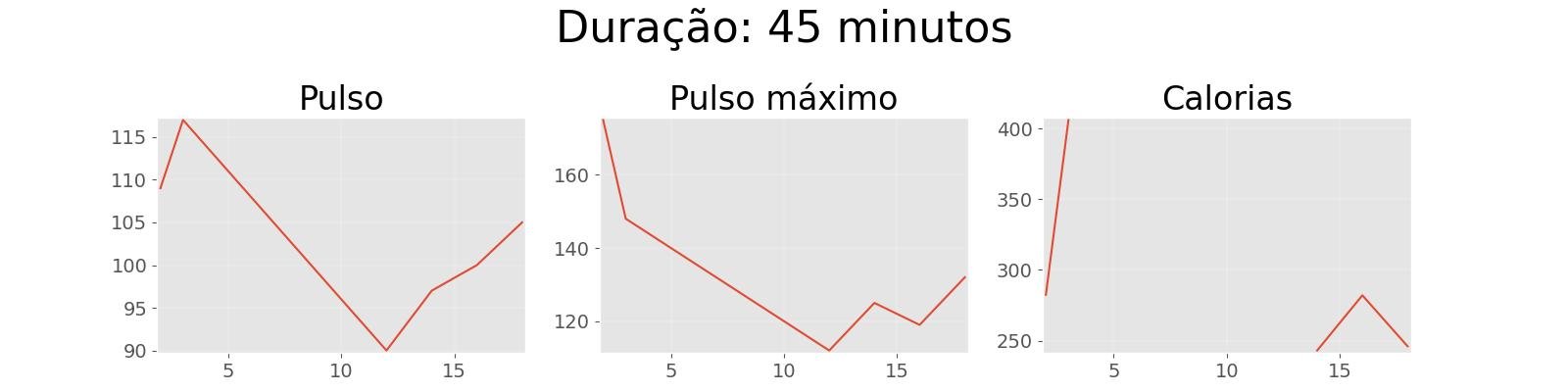
Seleção de valores únicos da coluna "Duracao".
df_duracao_unica = pd.unique(df['Duracao'])
print(df_duracao_unica)Gráficos de dispersão de pulsos das durações únicas:
for duracao in df_duracao_unica:
df_aux = df[df['Duracao'] == duracao]
fig, axes = plt.subplots(nrows=1, ncols=3, figsize=(16,4))
fig.suptitle('Duração: {} minutos'.format(duracao))
fig.tight_layout(pad=3.0)
fig.subplots_adjust(left=0.1, top=0.7, right=0.9, bottom=0.1, wspace=0.2, hspace=0.5)
df_aux['Pulso'].plot(ax=axes[0], title='Pulso')
df_aux['Pulsomax'].plot(ax=axes[1], title='Pulso máximo')
df_aux['Calorias'].plot(ax=axes[2], title='Calorias')
plt.show()